Los hongos establecen diferentes interacciones con las plantas. Cepas o especies de hongos estrechamente relacionadas entre sí pueden causar enfermedades o no, dependiendo de su repertorio de mecanismos de patogénesis y de la capacidad de la planta para defenderse. Los hongos patógenos atraen gran parte de nuestra atención porque causan importantes pérdidas en los cultivos o en los ecosistemas. Sin embargo, los hongos no patógenos también persisten en la naturaleza, y pueden sobrevivir en las superficies del huésped creciendo de manera epífita y estableciendo tipos específicos de interacciones con las plantas. La comparación de los genomas (repertorio de genes) y transcriptomas (genes expresados) de cepas patogénicas y no patogénicas en su interacción con la planta proporciona claves sobre los determinantes que conducen, o no, a la enfermedad.
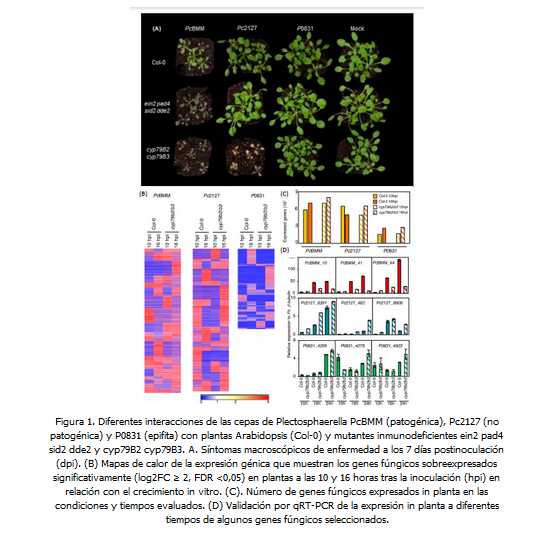
El hongo Plectosphaerella cucumerina es un patógeno de cultivos y un organismo modelo para el estudio de la resistencia basal y no huésped de Arabidopsis thaliana a hongos necrótrofos. En este trabajo, el grupo de “Inmunidad Innata de Plantas y Resistencia a Hongos Necrótrofos” del CBGP ha secuenciado, anotado y comparado por primera vez los genomas y transcriptomas de tres cepas del género Plectosphaerella con diferentes estilos de vida en A. thaliana: PcBMM, un patógeno natural de plantas silvestres Col-0, Pc2127, una cepa no patogénica para Col-0 pero patogénica en el mutante inmunodeprimido cyp79B3 cyp79B2, y P0831, una cepa no patogénica aislada de una población natural de A. thaliana y que crece epífiticamente en plantas Col-0 y cyp79B2 cyp79B3. Los genomas de estas cepas de Plectosphaerella son muy similares y no difieren en el número de genes con funciones relacionadas con la patogénesis, con la excepción de enzimas con actividad sobre carbohidratos (CAZymes) que son secretadas, que son hasta cinco veces más abundantes en la cepa patogénica PcBMM que en el resto. El análisis de los transcriptomas fúngicos en las etapas iniciales de plantas inoculadas con las cepas fungicas confirman el papel clave de las CAZymes secretadas en la interacción necrotrófica. Los investigadores del CBGP también han mostrado que el crecimiento epífito de P0831 en A. thaliana implica la transcripción de repertorios específicos de genes fúngicos. Estas son las primeras secuencias publicadas de genomas y transcritomasde Plectosphaerella spp., y también el primer proyecto de genómica de un hongo patógeno de plantas liderado por investigadores del CBGP, que han contado con la colaboración del grupo de Toni Gabaldón (CRG). Este estudio de genómica comparada puede contribuir a diseñar estrategias de protección de cultivos que actúen específicamente sobre hongos patógenos.
Publicación Original:
Muñoz-Barrios, A., Sopeña-Torres, S., Ramos, B., López, G., Del Hierro García, I., Díaz-González, S., González-Melendi, P., Mélida, H., Fernández-Calleja, V., Mixão, V., Martín Dacal, M., Marcet-Houben, M., Gabaldón, T., Sacristan, S., Molina, A. 2020. Differential expression of fungal genes determines the lifestyle of Plectosphaerella strains during Arabidopsis thaliana colonization. Molecular Plant-Microbe Interactions®. DOI: 10.1094/MPMI-03-20-0057-R