La publicación en Frontiers in Cell and Developmental Biology señala recursos de gran utilidad para toda la comunidad científica de investigadores en peces cebra, tanto en biología del desarrollo, fisiología, toxicología o biomedicina siendo posible su aplicación también en otros organismos modelo.
Un grupo de investigación del Centro Andaluz de Biología del Desarrollo (CABD) ha publicado un estudio en la revista Frontiers in Cell and Developmental Biology en el que señalan el desarrollo de una herramienta genética que facilita la generación de peces cebra para realizar estudios de expresión de genes con detalle en tejidos específicos.
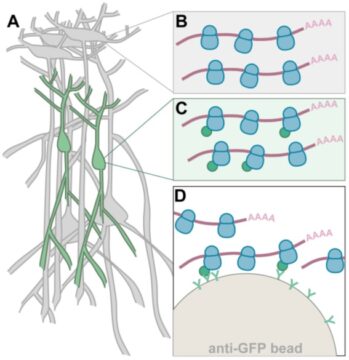
Hasta ahora se ha utilizado la técnica TRAP, Translating Ribosome Affinity Purification, que supone una gran ventaja para realizar análisis específicos de tejidos y ha sido utilizada en varios organismos como el pez cebra (Danio rerio) la mosca de la fruta (Drosophila melanogaster) o el ratón (Mus musculus). Sin embargo, para cada tipo de análisis es necesario crear un animal que contenga los ribosomas marcados con GFP, una molécula fluorescente, en el tejido objetivo. Esto supone una dificultad ya que la generación de este tipo de organismos es compleja y costosa en términos de tiempo y económicos. En este trabajo han desarrollado dos estrategias diferentes para facilitar la generación de peces cebra que sean aptos para el análisis TRAP. Ambas se basan en la introducción de la secuencia del ribosoma-GFP en el ADN, pero con algunas diferencias.
La primera herramienta, denominada trap-TRAP permite la generación aleatoria de un número indefinido de peces diferentes aptos para el análisis TRAP. Para ello, la secuencia del ribosoma-GFP se introduce aleatoriamente en el ADN. En lugar de dirigir su activación a un tejido específico se le añade un elemento que le permite imitar la dinámica de activación de los genes cercanos. Por ejemplo, si se inserta cerca de un gen específico de neuronas puede copiar su comportamiento dando lugar a neuronas con ribosomas fluorescentes.
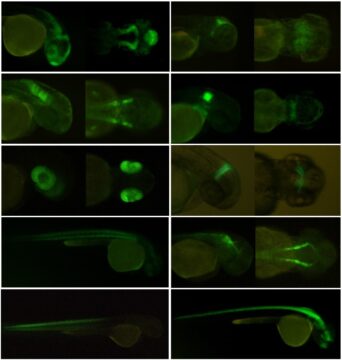
“Para probar esta herramienta desarrollamos un estudio piloto en el cual conseguimos obtener 53 líneas de peces con expresión ribosoma-GFP diferente, de los cuales 33 mostraban una expresión en tejidos muy específicos. Gracias a este estudio demostramos que con esta herramienta es posible generar líneas distintas de peces de una manera rápida, fácil y más económica. Además, también sería posible adaptar esta tecnología a otros organismos modelo”, afirma Juan Ramón Martínez Morales, investigador principal del estudio, del CABD.
“En la segunda estrategia utilizamos una herramienta ya existente que permite controlar la expresión de genes en tejidos concretos, denominada Gal4/UAS. Este sistema se basa en dos tipos diferentes de peces. En nuestro caso, los peces UAS contienen la secuencia ribosoma-GFP mientras que los peces Gal4 tienen la capacidad de activar la expresión de la secuencia contenida en el pez UAS en tejidos concretos. Para que esto ocurra solo hace falta cruzar los dos tipos de peces para que en la descendencia ambos elementos coincidan. De esta manera un mismo pez UAS ribosoma-GFP puede cruzarse con distintos peces Gal4 dando lugar a expresión en distintos tejidos. La combinación del sistema Gal4/UAS y la tecnología TRAP ya fue probada en Drosophila y nosotros lo hemos adaptado al pez cebra”, concluye el investigador del CABD.
En definitiva, estas dos herramientas facilitan la generación de peces para realizar estudios de expresión de genes en tejidos específicos. Estas herramientas genéticas abren un abanico de opciones para el estudio detallado de multitud de genes y sus funciones en diversos tejidos de un organismo vivo (en este caso en el pez cebra, se podría establecer fácilmente en gusanos y mosca de la fruta). Ambas son recursos de gran utilidad para toda la comunidad científica de investigadores en peces cebra, tanto en biología del desarrollo, fisiología, toxicología o biomedicina siendo posible su aplicación también en otros organismos modelo.
Una historia sobre genes contada desde el principio
La expresión de genes nos proporciona información muy útil sobre los procesos que están ocurriendo en un organismo, tejido o grupo de células. Pero “¿A qué nos referimos cuando hablamos de expresión de genes?”, se pregunta el coautor del estudio, Jorge Corbacho. Y nos explica: “la información genética de un individuo se encuentra almacenada en el ADN, moléculas que podemos imaginar como cadenas, compuestas de 4 tipos de eslabones simbolizados con las letras A, C, T y G. La información genética está codificada como una secuencia de esos 4 eslabones. Casi todas las células de un organismo poseen copias idénticas de las mismas cadenas de ADN. Sin embargo, no en todas se utiliza la misma porción de información. Según las necesidades o los procesos en los que se vea involucrada una célula, ésta requerirá que ciertos fragmentos de ADN, conocidos como genes, se activen y ejerzan su función. Por ejemplo, en una célula muscular se activarán genes específicos de células musculares, pero los genes que sean específicos de células neuronales permanecerán inactivos. Para que los genes ejerzan su función es necesario que se dé una sucesión de eventos. Generalmente, primero, la secuencia contenida en ellos es copiada en otro tipo de moléculas, conocidas como ARN. Las moléculas de ARN son más pequeñas y transportan la secuencia fuera del núcleo de la célula. Allí, puede dar lugar a la formación de proteínas gracias a la labor de unas máquinas celulares llamadas ribosomas. Los ribosomas leen las “instrucciones” contenidas en el ARN y a partir de ellas fabrican las proteínas. Las proteínas son las que en última instancia se encargan de realizar diversas funciones, como fabricar y mantener tejidos, llevar a cabo reacciones químicas, transportar sustancias o incluso permitir el movimiento de nuestros músculos”.
De esta forma, volviendo a la pregunta que nos hacíamos inicialmente, ¿a qué nos referimos cuando hablamos de expresión de genes? “Normalmente hacemos referencia al conjunto de genes activos en un tejido o tipo de célula en un momento concreto. Para poder identificarlos, existe una técnica que consiste en aislar las moléculas de ARN presentes en la célula y analizar sus secuencias. Como éstas son copias de los genes activos, esta técnica, denominada secuenciación de ARN o RNA-seq, puede proporcionarnos esta información. Estudiar la expresión de genes nos informa sobre los procesos que se están llevando a cabo en las células o tejidos, nos permite comprender más acerca de las bases moleculares de diversas enfermedades o entender el efecto que está teniendo la aplicación de un medicamento, entre otros ejemplos”. Pero no todo es sencillo y, a pesar de su gran utilidad esta técnica también comprende algunas limitaciones. “Por ejemplo, a veces queremos estudiar tejidos pequeños o que se encuentran muy unidos a otros. En esos casos, separar el tejido objetivo manualmente puede ser muy complicado, existiendo además el riesgo de mezclarlo con los adyacentes. Existen algunos métodos que utilizan máquinas para aislar las células en las que estamos interesados, pero normalmente estos métodos implican procesos que pueden estresarlas y alterar su perfil de expresión de genes.
Como alternativa, se desarrolló otra técnica conocida como TRAP (Translating Ribosome Affinity Purification). Esta técnica se basa en la obtención de ARN a partir de la purificación de ribosomas (recordemos que los ribosomas son las máquinas celulares que se encargan de fabricar proteínas a partir de la secuencia del ARN). Para poder ejercer su función, los ribosomas deben unirse al ARN, por lo que si conseguimos aislar los ribosomas podemos obtener las moléculas de ARN unidas a ellos.
Sobre el CABD
El CABD se fundó en el año 2003 como el primer instituto español especializado en el estudio de la Biología del Desarrollo. En 2017 el Departamento de Regulación Génica y Morfogénesis recibió la acreditación de Unidad de Excelencia María de Maeztu para el periodo 2017-2021 y ha sido ampliada para el CABD en su conjunto durante el periodo 2022-2025. El CABD, que se aloja en el edificio JA Campos Ortega, es un centro mixto cofinanciado por el Consejo Superior de Investigaciones Científicas (CSIC), la Junta de Andalucía y la Universidad Pablo de Olavide (UPO) de Sevilla.
El foco de investigación se ha escogido para acoger y promover a la prestigiosa escuela española de Biología del Desarrollo que se ha ido extendiendo por diferentes laboratorios internacionales. Actualmente el centro lo ocupan grupos jóvenes y dinámicos trabajando en desarrollo de ratón, pez cebra, Xenopus, Drosophila y Caenorhabditis. En el CABD también existen grupos estudiando control del ciclo celular en levaduras, regulación génica en bacterias y estrés oxidativo.
Referencia:
Jorge Corbacho, Estefanía Sanabria-Reinoso, Lorena Buono, Ana Fernández-Miñan and Juan R. Martínez-Morales. Trap-TRAP, a Versatile Tool for Tissue-Specific Translatomics in Zebrafish. (Enero 2022). Frontiers in Cell and Developmental Biology. DOI: 10.3389/fcell.2021.817191