El grupo de investigación en Genómica Computacional de la Universidad Pompeu Fabra ha descubierto nuevas alteraciones vinculadas a cáncer y ha puesto de relieve la importancia del transcriptoma en el desarrollo de tumores. Por primera vez, científicos estudian la variabilidad del transcriptoma celular en varios tumores a la vez, es decir, no se centran en el ADN, sino en los productos que se extraen de él. Los resultados del estudio aparecen en la revista Genome Research.
La información genética de las células se encuentra codificada en la cadena de ADN. Esta información es leída por la maquinaria celular, que primero generará el llamado ARN para después traducirlo a proteínas. Se trata de un proceso que requiere una regulación muy minuciosa. En ocasiones, el mismo gen puede dar lugar a distintas moléculas de ARN, las cuales producen diferentes proteínas, con funciones potencialmente muy diversas. Esto se debe al proceso conocido como “splicing alternativo”. Este splicing alternativo influye en la síntesis de la mayoría de las proteínas de las células eucariotas y su regulación depende de las proteínas de unión al ARN (RBPs, en sus siglas en inglés).
A pesar de que la relevancia del splicing alternativo en el funcionamiento celular es ampliamente conocida por la comunidad científica, el papel que juega en cáncer no ha hecho más que empezar a aflorar. Esto ha sido posible gracias a las nuevas técnicas de secuenciación y la disponibilidad de datos de secuencias de ARN de múltiples tumores a través de proyectos como el Atlas del Genoma de Cáncer (TCGA en sus siglas en inglés). Aunque el cáncer se origina a partir de mutaciones en el ADN, éstas tienen un impacto en el conjunto de moléculas de ARN de la célula, conocido como transcriptoma, que puede inducir y sostener mecanismos vinculados al desarrollo del cáncer. El equipo de investigación liderado por Eduardo Eyras, jefe del laboratorio de Genómica Computacional, ha descubierto ciertas alteraciones en las RBPs que generan cambios en el splicing alternativo vinculados con el desarrollo de cáncer.
Gracias a herramientas bioinformáticas diseñadas por el propio grupo de investigación, los científicos han podido realizar, en tiempo récord y usando recursos computacionales limitados, un análisis exhaustivo del ARN de más de 4.000 muestras tumorales de once tipos diferentes de cáncer extraídas del proyecto TCGA. Este análisis demuestra que las proteínas ligadas al ARN se encuentran con mucha frecuencia alteradas en los tumores humanos y que dichas alteraciones determinan el transcriptoma de las células e induce transformaciones celulares ligadas al desarrollo de cáncer. Dichas alteraciones habían permanecido invisibles hasta ahora a los métodos usados en grandes proyectos de análisis de genomas de cáncer.
En colaboración con los doctores Juan Valcárcel y Belén Miñana del Centro de Regulación Genómica (CRG), y los doctores Miguel Angel Pujana y Francesca Mateo del Instituto Catalán de Oncología (ICO), los autores han demostrado que introduciendo las alteraciones identificadas del transcriptoma en células no tumorales, éstas adquieren propiedades tumorales. Además de ampliar nuestros conocimientos sobre el papel de las RBPs en los tumores, los resultados destacan la importancia del splicing alternativo como mecanismo complementario en el desarrollo de cáncer, convirtiéndose en un nuevo factor a tener en cuenta en el estudio de esta enfermedad. Esta investigación abre nuevas vías para entender la biología del cáncer y buscar nuevas estrategias terapéuticas. Tal y como comentan los autores, “las alteraciones en el splicing alternativo son particularmente importantes en el contexto de tumores que no albergan mutaciones conocidas para las que se ha descrito una terapia y, por tanto, pueden abrir nuevas oportunidades para entender la biología del tumor y buscar nuevas terapias”.
Este trabajo ha sido posible gracias a la financiación de la Fundación Sandra Ibarra, del proyecto Consolider RNAREG, así como de proyectos del Plan Estatal y fondos FEDER.
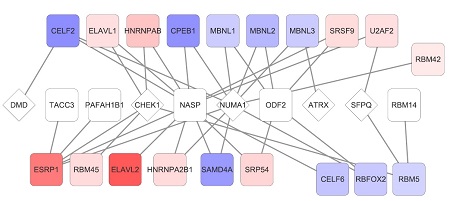
Figura: Red de regulación de splicing en genes del ciclo celular en cáncer de mama. Entre otros resultados, los autores han descrito la red de proteínas que interaccionan con el RNA (RBPs) que aumentan (rojo) o disminuyen (azul) su expresión en cáncer de mama, y controlan el splicing alternativo de genes relevantes para el ciclo celular. Algunos de estos genes se habían relacionado anteriormente con cáncer (rombos). En el caso de NUMA1, su papel en cáncer es aún desconocido. En este trabajo, los autores muestran que el splicing alternativo de NUMA1 induce propiedades tumorales en células normales de mama.
Trabajo de referencia: Endre Sebestyén, Babita Singh, Belén Miñana, et al. Large-scale analysis of genome and transcriptome alterations in multiple tumors unveils novel cancer-relevant splicing networks. Genome Research published online April 13, 2016. DOI:10.1101/gr.199935.115